1. SARS-CoV-2 Main Protease (Mpro)
1.1 Materiais e Métodos
Clonagem, expressão e purificação
A sequência codificante da enzima Mpro de SARS-CoV-2, incluindo a sequência de auto-clivagem, foi amplificada a partir do cDNA produzido a partir do RNA procedente de amostras de SARS-CoV-2 inativadas providas pelo grupo do Prof. Edison Durigon do ICB-USP e inserida no vetor pET_M11-LIC através do método LIC. O plasmídeo resultante foi utilizado para transformar a linhagem de expressão de E. coli BL21. As células contendo o plasmídeo foram crescidas a 37°C, 200 rpm em meio auto indutor ZYM 5052 até atingir DO600 de 0.6-0.8. A indução da expressão da proteína foi realizada reduzindo a temperatura para 18°C e crescendo as células por 16 h sob mesma agitação.
As células foram obtidas através de centrifugação (5000 x g, 40 min, 4°C) e em seguida ressuspendidas em tampão de lise (20 mM Tris pH 7.8, 150 mM NaCl, 1 mM DTT) e lisadas por sonicação (pulso ON 30s, pulso OFF 30s, 12 min). O extrato celular solúvel foi obtido por centrifugação (12000 x g, 30 min, 4°C). Devido à presença da sequência de reconhecimento da enzima, grande parte da proteína sofre o processo de auto-clivagem resultando na proteína em sua forma madura, já liberada de sua cauda de fusão 6xHIS tag. A pequena fração de enzima ainda com 6xHIS tag é removida do lisado utilizando resina contendo níquel (Qiagen). A proteína clivada foi purificada utilizando precipitação com sulfato de amônia. Foi adicionado 1.6 M sulfato de amônia no lisado celular sob agitação e incubado no gelo por 10 min. A proteína precipitada foi removida do extrato celular por centrifugação (12000 x g, 30 min, 4°C). A proteína foi então ressuspendida em tampão de gel filtração contendo 20 mM Tris pH 7.8, 150 mM NaCl, 1 mM EDTA, 1 mM DTT e purificada por exclusão de tamanho molecular utilizando coluna HiLoad 26/100 Superdex 200 (GE Healthcare) pré-equilibrada em tampão de gel filtração. O tampão foi trocado para 20 mM Tris pH 8.0, 1 mM DTT e então foi realizado o útimo passo de purificação através de troca iônica em coluna Mono-Q 5/50 GL (GE Healthcare), na qual a proteína de interesse foi eluida com gradiente de tampão B (20 mM Tris pH 8.0, 1 M NaCl, 1 mM DTT). Ao final, o tampão foi novamente trocado para 20 mM Tris pH 7.8, 150 mM NaCl, 1 mM EDTA, 1 mM DTT. A pureza foi analisada por SDS-PAGE e a concentração determinada usando a absorbância medida em 280 nm e seu coeficiente de extinção teórico de 32890 M-1.cm-1.
Ensaios de atividade e inibição
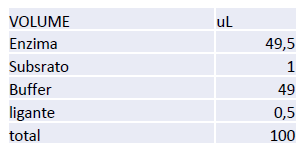
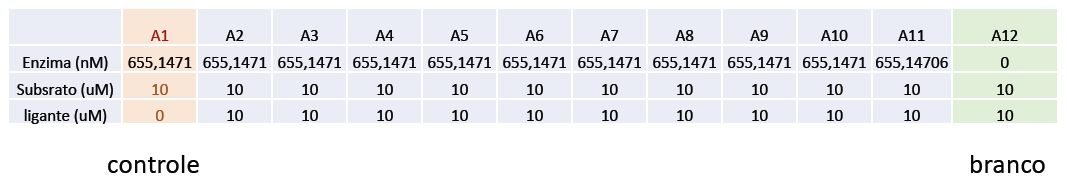
Os ensaios de atividade foram realizados de acordo com o método descrito. A enzima foi diluída para uma concentração final de 40 nM em tampão de reação (20 mM Tris, 1 mM DTT, 1 mM EDTA, pH 7.3) e incubada por 10 minutos a 37°C. A reação foi iniciada com adição de 10 µM de substrato Dabcyl-KTSAVLQ↓SGFRKM-E(Edans)-NH2. A reação foi mantida a temperatura constante a 37°C, com a fluorescência sendo monitorada a cada 30 segundos durante 60 minutos utilizando o equipamento SpectraMax Gemini EM Microplate Reader (Molecular Devices). As reações foram inicialmente feitas em placas Corning® brancas de 96 poços e posteriormente escalonadas para placas de 384 poços. Os comprimentos de onda utilizados para excitação e emissão foram respectivamente 360 nm e 460 nm.
Para os ensaios de inibição, foi adicionado 10 µM de composto (1% DMSO) na reação contendo enzima, e o ensaio foi conduzido da mesma maneira descrita acima. A porcentagem de inibição dos compostos foi calculada a partir de seus valores de fluorescência no tempo de 10 min com relação a uma reação controle (1% DMSO). Foram testados todos os compostos disponíveis no laboratório, totalizando um total de aproximadamente 4.000 moléculas. Dentre eles estão as séries de peptídeos sintéticos sintetizadas e fornecidas em colaboração com o Prof. Eduardo Maffud Cilli (Universidade Estadual Paulista Julio de Mesquita Filho – UNESP, Araraquara, Brasil), as séries Pandemic Response Box (400 compostos), Pathogen Box (400 compostos) e Malaria Box (400 compostos) provenientes da organização Medicines for Malaria Venture (MMV) e a biblioteca de compostos proveniente da colaboração com a empresa farmacêutica Collaborations Phamaceuticals, Inc., Carolina do Norte, EUA. (2.600 compostos), denominada série CPIs.
2 SARS-CoV-2 Papain-like Protease (PLpro)
2.1 Materiais e Métodos
Clonagem e expressão em larga escala
A sequência codificante da proteína PLpro de SARS-CoV-2 foi amplificada a partir do cDNA produzido a partir do RNA procedente de amostras de SARS-CoV-2 inativadas providas pelo grupo do Prof. Edison Durigon do ICB-USP. O produto da amplificação foi enviado ao grupo do Prof. Flávio H. da Silva (Universidade Federal de São Carlos – UFSCar, São Carlos – São Paulo, Brasil) onde foi clonado em vetor de expressão pET-28a e gentilmente disponibilizado ao nosso grupo. Essa construção contém uma cauda de 6xHis na região C-terminal. O vetor foi transformado na linhagem de E. coli BL21, seguida da expressão em larga escala em meio LB, onde as culturas bacterianas foram crescidas sob agitação a 37 °C até atingir DO de 0.6. Na sequência, a expressão foi induzida pela adição de 0.5 mM de IPTG e 1 mM de sulfato de zinco (ZnSO4) e após a indução, as culturas foram mantidas a 18 °C por 20 h. Ao final do período de expressão, as células foram separadas por centrifugação e ressuspendidas em tampão de lise (50 mM Tris-HCl pH 8.0, 150 mM NaCl, 10 mM Imidazol e 1 mM de DTT).
Purificação da enzima
A lise celular foi realizada com adição de 250 µg/mL de lisozima e 10 U/mL de benzonase (Cellco Biotec), seguida de sonicação. Em seguida, o lisado celular obtido foi centrifugado (15000 x g, 40 min, 4°C) para a obtenção da fração solúvel. A primeira etapa de purificação foi realizada por cromatografia de afinidade a metal imobilizado (IMAC) utilizando a coluna Histrap HP (GE Healthcare). Após a lavagem, a proteína foi eluida com tampão contendo 50 mM Tris-HCl, pH 8.5, 150 mM NaCl, 250 mM Imidazol e 1 mM DTT. Em seguida, foi realizada uma segunda etapa de purificação por cromatografia de exclusão molecular (gel filtração) utilizando a coluna Superdex 75 16/60 (GE Healthcare) previamente equilibrada com tampão 20 mM Tris-HCl pH 7.4, 100 mM NaCl e 1 mM TCEP. A pureza da amostra foi analisada por SDS-PAGE 12.5%. A proteína foi concentrada a 1.0 mg/mL e armazenada para a realização dos ensaios de atividade enzimática. A concentração da amostra foi determinada utilizando Nanodrop 1000 spectrophotometer.
Ensaios de inibição da atividade enzimática
O ensaio de atividade enzimática da PLpro foi padronizado utilizando diferentes concentrações de enzima e do substrato enzimático fluorescente, a fim de obter as condições adequadas de detecção de intensidade de fluorescência para o ensaio. O substrato enzimático fluorescente utilizado foi o Abz-TLKGG//APIKQ-EDDnp, que foi sintetizado e gentilmente disponibilizado pela Profa. Maria Aparecida Juliano (Universidade Federal de São Paulo – UNIFESP, São Paulo, Brasil). Ao final, foi estabelecida a concentração final da enzima de 70 nM e o substrato na concentração final de 27 µM. O ensaio foi inicialmente realizado em microplacas brancas de 96 poços (Corning) e posteriormente escalonado para placas de 384 poços. A proteína foi diluída em tampão de ensaio (50 mM HEPES pH 7.5, 0.01 % Triton e 5 mM DTT) e incubada com 10 µM de cada composto ou peptídeo, a 37 °C por 30 min. Após isso, o substrato diluído em tampão de ensaio foi adicionado e a atividade enzimática mensurada em espectrofluorímetro Spectramax Gemini EM (Molecular devices), com λex= 320 nm e λem= 420 nm, a 37 °C durante 30 min.
Os compostos ou peptídeos que apresentaram percentual de inibição da atividade enzimática de no mínimo 80% foram selecionados para o ensaio seguinte de dose-resposta para determinação de seus IC50.
3 SARS-CoV-2 NendoU (nsp15)
3.1 Materiais e Métodos
Clonagem, expressão e purificação da enzima
O gene referente à proteína não estrutural endoribonuclease NendoU (NSP15) foi amplificado por RT-PCR a partir do RNA extraído do vírus isolado do paciente 01 brasileiro de COVID-19 (GenBank: QIG55993.1), gentilmente cedido pelo Prof. Edson Durigon, clonado em vetor pET-M11 e transformado em células de expressão BL21(DE3). Após testes de expressão as condições de expressão e purificação puderam ser otimizadas.
As células BL21(DE3) transformadas foram cultivadas em meio LB Lennox suplementado com canamicina a 37 °C até DO600 = 1,0, e mantidas por 16 h a 18 °C após a indução com 0,5 mM de isopropil β-D-1-tiogalactopiranosídeo. As células colhidas foram ressuspensas em Tampão A (50 mM TRIS-HCl pH 8,0, 300 mM NaCl, 10% de glicerol, 20 mM de imidazol) suplementado com 0,1 mg mL-1 de lisozima, 10 U de benzonase (Cellco Biotec) e 1,0 mM DTT, e interrompido usando sonicação. O lisado limpo foi passado através de uma HisTrap HP 5 mL (GE Healthcare) equilibrada em Tampão A, e a proteína foi eluída com Tampão A suplementado com 300 mM imidazol. O imidazol excessivo foi removido usando uma coluna Sephadex XK 26/60 (GE Healthcare) equilibrada com tampão A. Para obter NSP15 na forma monomérica (NendoUmon), a amostra foi incubada durante a noite a 8 °C com DTT 4 mM e protease de TEV 0,1 mg mL-1, depois passada através de uma HisTrap HP de 5 mL equilibrada em Tampão A para remover a proteína não clivada e a protease de TEV. A última etapa de purificação foi realizada por cromatografia de exclusão de tamanho usando uma coluna HiLoad Superdex 75 16/60 (GE Healthcare) equilibrada com 20 mM HEPES pH 7,5, 150 mM NaCl, 5% glicerol (v / v), 0,5 mM TCEP. A pureza foi confirmada por SDS-PAGE 12%. A concentração de proteína foi estimada usando o coeficiente de extinção teórico a 280 nm de 33.140 M-1 cm-1.
Ensaio de atividade enzimática
Os ensaios de inibição foram realizados com a enzima no estado monomérico NendoUmon a uma concentração de 105 nM, utilizando como substrato o oligonucleotídeo fluorogênico 5’6-FAM-dArUdAdA-6-TAMRA3’ (Genscript). As reações foram realizadas em tampão 50 mM Bistris pH 6,0 e monitoradas por 120 min no equipamento Stratagene Mx3005P (Agilent Technologies) utilizando filtros FAM, à 25 oC. Primeiramente, um ensaio de ponto único em estratégia HTS foi realizado com os compostos à 20 µM e a atividade relativa foi calculada a partir do coeficiente angular em os tempos de reação 5 e 90 min. Potenciais alvos que apresentaram inibição de no mínimo 80% foram selecionados para os ensaios de concentração resposta (IC50). Para os ensaios de IC50, cada composto foi diluído serialmente de 20 a 0,6 µM.
4. Publicações deste projeto referentes à biologia estrutural e triagens virtual, enzimática,
cristalográfica:
(1) “A Crystallographic Snapshot of SARS-CoV-2 Main Protease Maturation Process”.
G. D. Noske, A. M. Nakamura, V. O. Gawriljuk, R. S. Fernandes, G. M. A. Lima, H. V. D. Rosa,
H. D. Pereira, A. C. M. Zeri, A. F. Z. Nascimento, M. C. L. C. Freire, G. Oliva1, A. S. Godoy.
bioRxiv 2020.12.23.424149; doi: https://doi.org/10.1101/2020.12.23.424149
Submitted to Proc. Natl. Acad. Sci. USA
(2) “Non-toxic dimeric peptides derived from the Bothropstoxin-I are potent SARS-CoV-2 and
Papain-Like Protease inhibitors”
Marjorie C. L. C. Freire, Gabriela D. Noske, Paulo R. S. Sanches, Norival A. Santos-Filho,
Victor G. F. Oliveira, Eduardo P. de Souza, Victor H. N. Rabesquine, Mariana O. de Godoy,
Aline M. Nakamura, Rafaela S. Fernandes, Andre S. Godoy, Maria A. Juliano, Bianca M.
Peres, Cecília Gomes Barbosa, Carolina B. Moraes, Lucio H. Freitas-Junior, Eduardo M.
Cilli, Rafael V. C. Guido, Glaucius Oliva.
Submitted to Molecules
5 Referencias relativas à biologia estrutural e ensaios enzimáticos
- Ma, C. et al. Boceprevir, GC-376, and calpain inhibitors II, XII inhibit SARS-CoV-2 viral replication by targeting the viral main protease. Cell Res. 30, 678–692 (2020).
- Fu, L. et al. Both Boceprevir and GC376 efficaciously inhibit SARS-CoV-2 by targeting its main protease. Nat. Commun. 11, 1–8 (2020).
- Ferreira, R. da S. et al. Crystal Structure of Crataeva tapia Bark Protein (CrataBL) and Its Effect in Human Prostate Cancer Cell Lines. PLoS One 8, e64426 (2013).
- Hansen, D. et al. Crystal structure of a novel cysteinless plant Kunitz-type protease inhibitor. Biochem. Biophys. Res. Commun. 360, 735–740 (2007).
- Ulian Araújo, A. P. et al. Kunitz-type Bauhinia bauhinioides inhibitors devoid of disulfide bridges: Isolation of the cDNAs, heterologous expression and structural studies. Biol. Chem. 386, 561–568 (2005).
- Ghosh, A. K. et al. Severe acute respiratory syndrome coronavirus papain-like novel protease inhibitors: Design, synthesis, protein-ligand X-ray structure and biological evaluation. J. Med. Chem. 53, 4968–4979 (2010).
- Bakovic, A. et al. Brilacidin, a COVID-19 Drug Candidate, Exhibits Potent In Vitro Antiviral Activity Against SARS-CoV-2. bioRxiv 2020.10.29.352450 (2020). doi:10.1101/2020.10.29.352450
- Nogueira Batista, M. et al. GA-Hecate antiviral properties on HCV whole cycle represent a new antiviral class and open the door for the development of broad spectrum antivirals OPEN. doi:10.1038/s41598-018-32176-w
- Costa, T. R. et al. Myotoxic phospholipases A2 isolated from Bothrops brazili snake venom and synthetic peptides derived from their C-terminal region: Cytotoxic effect on microorganism and tumor cells. Peptides 29, 1645–1656 (2008).
- Santos-Filho, N. A. et al. Synthesis and characterization of an antibacterial and non-toxic dimeric peptide derived from the C-terminal region of Bothropstoxin-I. Toxicon 103, 160–168 (2015).
- Lomonte, B., Profile, S., Angulo, Y. & Moreno, E. Synthetic Peptides Derived from the C-Terminal Region of Lys49 Phospholipase A2 Homologues from Viperidae Snake Venoms: Biomimetic Activities and Potential Applications Synthetic Peptides Derived from the C-Terminal Region of Lys49 Phospholipase A 2 Homologues from Viperidae Snake Venoms: Biomimetic Activities and Potential Applications. Curr. Pharm. Des. 16, 3224–3230 (2010).
- De Palma, A. M. et al. Mutations in the nonstructural protein 3A confer resistance to the novel enterovirus replication inhibitor TTP-8307. Antimicrob. Agents Chemother. 53, 1850–1857 (2009).
- Albulescu, L. et al. Uncovering oxysterol-binding protein (OSBP) as a target of the anti-enteroviral compound TTP-8307. Antiviral Res. 140, 37–44 (2017).
- Vonrhein, C. et al. Data processing and analysis with the autoPROC toolbox. Acta Crystallogr. Sect. D Biol. Crystallogr. 67, 293–302 (2011).
- Winter, G. et al. DIALS: Implementation and evaluation of a new integration package. Acta Crystallogr. Sect. D Struct. Biol. 74, 85–97 (2018).
- Kabsch, W. XDS. Acta Crystallogr D Biol Crystallogr 66, 125–132 (2010).
- Krojer, T. et al. The XChemExplorer graphical workflow tool for routine or large-scale protein-ligand structure determination. Acta Crystallogr. Sect. D Struct. Biol. 73, 267–278 (2017).
- Potterton, E., Briggs, P., Turkenburg, M., Dodson, E. & IUCr. A graphical user interface to the CCP 4 program suite. Acta Crystallogr. Sect. D Biol. Crystallogr. 59, 1131–1137 (2003).
- Adams, P. D. et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr. Sect. D 66, 213–221 (2010).
- McCoy, A. J. et al. Phaser crystallographic software. J Appl Crystallogr. 40, 658–674 (2007).
- Pearce, N. M. et al. A multi-crystal method for extracting obscured crystallographic states from conventionally uninterpretable electron density. Nat. Commun. 8, 1–8 (2017).
- Emsley, P., Lohkamp, B., Scott, W. G. & Cowtan, K. Features and development of Coot. Acta Crystallogr D Biol Crystallogr 66, 486–501 (2010).
- Smart, O. S. et al. Exploiting structure similarity in refinement: Automated NCS and target-structure restraints in BUSTER. Acta Crystallogr. Sect. D Biol. Crystallogr. 68, 368–380 (2012).
- Chen, V. B. et al. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Crystallogr D Biol Crystallogr 66, 12–21 (2010).
- Chiu, P. L. et al. Evaluation of super-resolution performance of the K2 electron-counting camera using 2D crystals of aquaporin-0. J. Struct. Biol. 192, 163–173 (2015).
- Zheng, S. Q. et al. MotionCor2: Anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods 14, 331–332 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A. CryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination. Nat. Methods 14, 290–296 (2017).
- Scheres, S. H. W. RELION: Implementation of a Bayesian approach to cryo-EM structure determination. J. Struct. Biol. 180, 519–530 (2012).
- Scheres, S. H. W. & Chen, S. Prevention of overfitting in cryo-EM structure determination. Nature Methods 9, 853–854 (2012).
- Afonine, P. V. et al. Real-space refinement in PHENIX for cryo-EM and crystallography. Acta Crystallogr. Sect. D Struct. Biol. 74, 531–544 (2018).
Detalhes para envio do composto:
– Composto à 20 mM, diluído em 100% DMSO
– Mínimo de 10 uL
– Composto será testado em ponto único à 10 uM
– IC50 será determinado para compostos que inibirem 80% da atividade
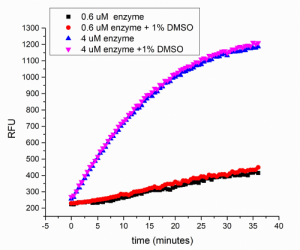
Resultados esperados
– Inibição em ponto único à 10 uM
– Z-score da placa