1 Metodologia e padronização do Ensaio Fenotípico SARS-CoV-2 x células Vero E6 em plataforma HCS
1.1 Laboratório de Nível de Segurança 3
Todos os procedimentos envolvendo o vírus SARS-CoV-2 são realizados no laboratório de biossegurança nível 3 (NB3) do Instituto de Ciências Biomédicas da Universidade de São Paulo. Para isso a equipe de pesquisa realizou um curso teórico, seguido de treinamento técnico e avaliação. Somente após a aprovação no treinamento a equipe deu início à rotina de trabalho no laboratório NB3.
1.2 SARS-CoV-2: amplificação e titulação viral
O SARS-CoV-2 utilizado nos ensaios fenotípicos (HIAE-02: SARS-CoV-2/ SP02/humano/2020/BRA, GenBank No. de Acesso MT126808.1) foi isolado de uma amostra nasofaríngea de um paciente confirmado de Covid-19 no Hospital Israelita Albert Einstein, São Paulo, SP, Brasil. O estoque de SARS-CoV-2 foi mantido em baixa passagem.
Para a amplificação viral, inicialmente as células Vero E6 (ATTC) foram cultivadas e mantidas em DMEM alta glicose (Sigma-Aldrich) suplementado com 10% de soro bovino fetal (SBF) inativado pelo calor (Thermo Scientific) e 100 U/mL de penicilina e 100 μg/mL de Estreptomicina (Thermo Scientific), a 37 °C em 5% de CO2. Após atingirem 80 a 90% de confluência, o meio de cultura foi removido, as células foram lavadas em meio sem SFB e em seguida foram infectadas com SARS-CoV-2. Após 1 hora de adsorção, as células foram mantidas em meio DMEM de alta glicose (Sigma-Aldrich), suplementado com 2% de SFB. Após 2 a 3 dias de infecção, o sobrenadante das células infectadas foi coletado, centrifugado para remoção de debris e armazenado em alíquotas a – 80 °C.
O título viral foi determinado pelo método Plaque Assay, modificado de Mendoza et. al. (2020). Resumidamente, o método aqui utilizado permite quantificar o número de partículas virais viáveis a partir da determinação das Unidades Formadoras de Placa (Plaque forming units, PFU) em volume de sobrenadante de cultura contendo vírus. Resumidamente, 1×105 células Vero CCL-81 (ATCC) foram semeadas em cada poço de uma placa de 24 poços em DMEM alta glicose suplementado com 10% de SBF como descrito anteriormente, a 37 °C em 5% de CO2. Após 24 h, a suspensão coletada de culturas de tecido infectadas com SARS-CoV-2 foi descongelado, homogeneizado e submetido a diluição seriada em fator 1:10 (v/v) em, 1 mL de meio de cultura DMEM alta glicose. Em seguida, o meio de cultura foi removido da placa de 24 poços e 400 μL de meio com diluições em série de SARS-CoV-2 foram adicionados a cada poço. A placa foi então incubada por 1 h a 37 °C com 5% de CO2, para adsorção viral. Em seguida, o meio foi novamente removido dos poços e substituído por 500 μL de DMEM alta glicose suplementado como acima, adicionado 2-3% de carboximetilcelulose. A placa foi incubada por 72 h, a 37 ˚C em atmosfera umidificada contendo 5% de CO2. Após esse período, o meio foi removido, a placa foi fixada com paraformaldeído 4% em PBS (m/v) pH 7,4 por 15 min e corada com violeta cristal 1% em etanol 10% (m/v/v). O número de plaques foi avaliado visualmente, contado e o título do vírus foi calculado como unidades formadoras de placa (PFU)/mL.
O método TCID50/ml (50% tissue culture infectious dose assay) foi padronizado em paralelo com a titulação pelo método Plaque Assay, mostrando-se um método alternativo de titulação. Células Vero CCL-81 (2×104 células/poço) foram plaqueadas em placas de 96 poços, 24 horas antes do experimento. O vírus foi diluído em série em meio e transferido para a placa de 96 poços, após 1 hora de adsorção viral, foi adicionado meio DMEM de alta glicose (Sigma-Aldrich) conforme especificações anteriores, suplementado com 2% de soro bovino fetal, sendo a placa foi incubada por 72 horas. Após tal período, as monocamadas foram fixadas e coradas com Naphthol Blue Black (Sigma-Aldrich) dissolvido em acetato de sódio-ácido acético. O título viral foi determinado e expresso em TCID 50/mL usando o algoritmo de Spearman & Kärber, conforme descrito por Hierholzer et al. (1996).
1.3. Ensaio fenotípico para descoberta de antivirais para SARS-CoV-2
O ensaio é realizado em placas de 384 poços (Greiner Bio-One), conforme procedimento detalhado por Sales-Medina et.al., 2020. Inicialmente, 2000 células Vero E6 foram semeadas em cada poço, em 40 µL de meio DMEM de alta glicose (Sigma-Aldrich) suplementado com 10% de SFB. As placas foram incubadas a 37 °C/5% de CO2.
Paralelamente, os compostos foram preparados para triagem. Para triagem primária, os compostos foram dissolvidos em DMSO na concentração de 2 mM para compostos sintéticos ou purificados ou 2 mg/mL para produtos naturais complexos (extratos e frações). Para triagens confirmatórias, os compostos foram dissolvidos em DMSO na concentração de 10 a 20 mM ou então 10 a 20 mg/mL. Para ensaios confirmatórios, os compostos foram então diluídos serialmente em DMSO na proporção de 1:2 (v/v), em dez pontos diferentes. O composto cloroquina foi preparado da mesma forma como controle positivo para atividade antiviral in vitro. Os compostos foram plaqueados em placas de polipropileno (Greiner Bio-One) e congelados a -20 ˚C.
No dia seguinte, as placas de compostos foram descongeladas e os compostos foram diluídos 33,33x (v/v) em PBS em uma segunda placa (placa intermediária). Em paralelo, o sobrenadante foi removido dos poços da placa contendo a cultura de Vero E6, aos quais foram acrescentados meio fresco sem SFB para um volume de 30 µL, além de 10 µL da solução de compostos em PBS da placa intermediária.
Em um laboratório NB3, partículas virais SARS-CoV-2 foram adicionadas às células em 10 μL de DMEM Alta glicose por poço na multiplicidade de infecção de 0,05 a 0,1. Células infectadas tratadas com DMSO 0,5% (veículo), células infectadas tratadas com cloroquina 25 µM (controle positivo) e células não infectadas tratadas com DMSO 0,5% são utilizadas como controles. A placa de ensaio é então incubada por 1 h a 37 ˚C/5% de CO2 para a adsorção viral, seguida pela adição de 10 μL de DMEM com alto teor de glicose suplementado com 6% de soro bovino fetal por poço. As placas foram incubadas por 32 h adicionais a a 37 ˚C/5% de CO2 e em seguida foram fixadas com paraformaldeído 4% (v/v) em PBS pH 7,4, e submetidas ao ensaio de imunofluorescência indireta para determinação da infecção celular por SARS-CoV-2. Brevemente, as células infectadas e fixadas foram lavadas com PBS, bloqueadas em albumina sérica bovina (BSA) 4% em PBS (v/v) pH 7,4 (BSA-PBS), sequencialmente incubadas com anticorpos primário e secundário, lavadas novamente em PBS, e por fim coradas com DAPI 5 µg/mL (Sigma-Aldrich) para marcação dos núcleos das células, conforme descrito (Sales-Medina et.al., 2020). Como anticorpo primário, foram utilizados um anticorpo policlonal de coelho anti-proteína de nucleocapsídeo de SARS-CoV-2 (GeneTex) a 2 µg/mL em BSA-PBS, ou soro humano de paciente conlavescente de covid-19 diluído 1:1000 (v/v) em BSA-PBS, ou soro de cavalo anti-proteína S de SARS-CoV-2 (gentilmente cedido pelo Prof. Jerson Lima Silva, UFRJ) diluído 1:1000 (v/v) em BSA-PBS. Como anticorpo secundários, foram utilizados anticorpo de cabra anti-IgG de coelho conjugado Alexa 488 (Thermo Scientific), anticorpo de cabra anti-IgG humana conjugado a Alexa 488 (Thermo Scientific), diluídos a 0.5 µg/mL em PBS, ou anticorpo de cabra anti-IgG de cavalo conjugado a DyLight 488 (Bethyl Labs), diluído a 0,25 µg/mL em PBS.
As placas são então submetidas a imageamento em um equipamento de High Content Screening Operetta (Perkin Elmer) utilizando uma objetiva de magnificação de 20´ e filtros apropriados para fluorescências azul (DAPI) e verde (Alexa ou DyLight 488). Foram adquiridas 9 imagens por poço. As imagens adquiridas são analisadas no software Harmony (Perkin Elmer), versão 3.5.2. A análise das imagens consistiu na identificação e contagem de células Vero E6 com base na segmentação nuclear, e infecção viral com base na coloração citoplasmática detectada pelo ensaio de imunofluorescência. A taxa de infecção (IR) é calculada como a razão entre o número de células infectadas e o número de células totais contadas em cada poço. A taxa de sobrevivência celular é calculada como o número de células contadas em cada poço dividido pelo número médio de células nos poços de controle negativo (células infectadas tratadas com veículo, DMSO 0,5%), multiplicado por 100. A atividade antiviral é determinada pela normalização da IR ao controle positivo (células não infectadas tratadas com DMSO 0,5%) e ao controle negativo.
Os ensaios de ponto único são realizados em duas replicatas independentes e os ensaios de curva de dose resposta são realizados em três replicatas independentes. Em triagens primárias, as análises da atividade antiviral e de sobrevivência celular (ou atividade citotóxica do composto sobre células infectadas) são determinadas a partir da média dos valores obtidos em duplicatas. Em ensaios confirmatórios, a atividade dos compostos é determinada utilizando o software GraphPad Prism versão 7.0 (GraphPad Software). Os valores de atividade antiviral e sobrevivência celular são plotados em tabelas do tipo XY e, após a transformação das concentrações exponenciais para escala log, os dados são ajustados em uma curva sigmoidal dose-resposta de slope variável. As concentrações equivalentes a EC50 e a CC50, que são, respectivamente, as concentrações que reduzem a infecção viral em 50% em relação aos controles e a concentração que reduz a população de células em 50% em relação ao controle negativo, foram determinadas por interpolação a partir das curvas sigmoides ajustadas aos dados.
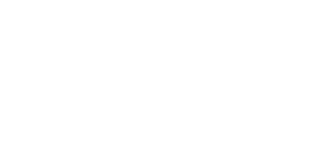
Até que se chegasse a um protocolo final, foram testadas diferentes densidades celulares e diferentes multiplicidades de infecção, em diferentes tempos de incubação, com detecção da infecção utilizando anticorpos obtidos comercialmente, e soros obtidos de doador humano (paciente de covid-19 convalescente) e de produção de soro terapêutico de cavalos imunizados com proteína S recombinante (dados não mostrados). A Figura 1 esquematiza o ensaio de triagem fenotípica em seu formato final, conforme descrito acima. Na Fig. 1A, o esquema geral do ensaio e do tipo de atividade observada para os compostos testados. Em Fig. 1B, imagens representativas da análise de High Content Screening para quantificação da atividade antiviral dos compostos e da sobrevivência celular. A metodologia de ensaio fenotípica desenvolvida foi publicada em formato preprint na plataforma Biorxiv (Sales-Medina et.al., 2020)
Durante a fase de desenvolvimento de ensaio fenotípico, observou-se uma grande variabilidade nas taxas de infecção de células Vero E6 por SARS-CoV-2, em diferentes poços de uma mesma placa e também entre diferentes placas (dados não mostrados). A variabilidade natural da infecção se manteve mesmo em condições de infecção mais altas, com multiplicidades de infecção maiores, como 1 e 0,5 partículas virais por célula (dados não mostrados), e por isso optou-se em manter uma MOI de 0,1. Determinou-se também que em tempo de ensaio de infecção de 32 a 34 horas seria mais adequado para imagear as células antes que o efeito citopático causado pelo SARS-CoV-2 (dados não mostrados).
1.4. Manuscrito publicado em plataforma de acesso aberto
Douglas Ferreira Sales-Medina, Ludmila Rodrigues Pinto Ferreira, Lavínia M. D. Romera, Karolina Ribeiro Gonçalves, Rafael V. C. Guido, Gilles Courtemanche, Marcos S. Buckeridge, Édison L. Durigon, Carolina B. Moraes, Lucio H. Freitas-Junior. Discovery of clinically approved drugs capable of inhibiting SARS-CoV-2 in vitro infection using a phenotypic screening strategy and network-analysis to predict their potential to treat covid-19. bioRxiv 2020.07.09.196337; doi: https://doi.org/10.1101/2020.07.09.196337
1.5. Referencias relativas aos ensaios fenotípicos
- Mendoza, E. J., Manguiat, K., Wood, H., & Drebot, M. (2020). Two detailed plaque assay protocols for the quantification of infectious SARS-CoV-2. Current Protocols in Microbiology, 57, e105. doi: 10.1002/cpmc.105
- Hierholzer JC, Killington RA. Virus isolation and quantitation. Virology Methods Manual. 1996;25-46. doi:10.1016/B978-012465330-6/50003-8.
- Douglas Ferreira Sales-Medina, Ludmila Rodrigues Pinto Ferreira, Lavínia M. D. Romera, Karolina Ribeiro Gonçalves, Rafael V. C. Guido, Gilles Courtemanche, Marcos S. Buckeridge, Édison L. Durigon, Carolina B. Moraes, Lucio H. Freitas-Junior. Discovery of clinically approved drugs capable of inhibiting SARS-CoV-2 in vitro infection using a phenotypic screening strategy and network-analysis to predict their potential to treat covid-19. bioRxiv 2020.07.09.196337; doi: https://doi.org/10.1101/2020.07.09.196337
- Takayama, K. In vitro and Animal Models for SARS-CoV-2 research. Trends Pharmacol Sci 41, 513–517 (2020).
- Hoffmann, M. et al. Chloroquine does not inhibit infection of human lung cells with SARS-CoV-2. Nature 1–5 (2020) doi:10.1038/s41586-020-2575-3.
- Mellott, D. M. et al. A cysteine protease inhibitor blocks SARS-CoV-2 infection of human and monkey cells. Biorxiv 2020.10.23.347534 (2020) doi:10.1101/2020.10.23.347534.
Compound plates:
1. Prepare an Excel form with the following information:
a. Date of shipment
b. Supplier
c. Compound ID (codes are fine, no need to include compound structure)
d. Volume/well
e. Compound concentration
f. Plate map/layout
g. Specify disease model(s) to be screened/tested
2. Plate specification:
a. Format:
A. Polypropylene plates with conical well bottom, regular SBS formats.
Suggestions: Greiner Small Volume, Deep Well Plates, Cat. No. 784 201 (384
wells) or Greiner Masterblock 0.5 ml Plates, Cat. No. 786201 (96 wells)
B. Specify the position of empty wells for controls.
Preferred: columns 1, 2, 23 and 24 (384 well plates) or columns 1 and 12
(96 well plaltes) left empty for control addition (as shown):
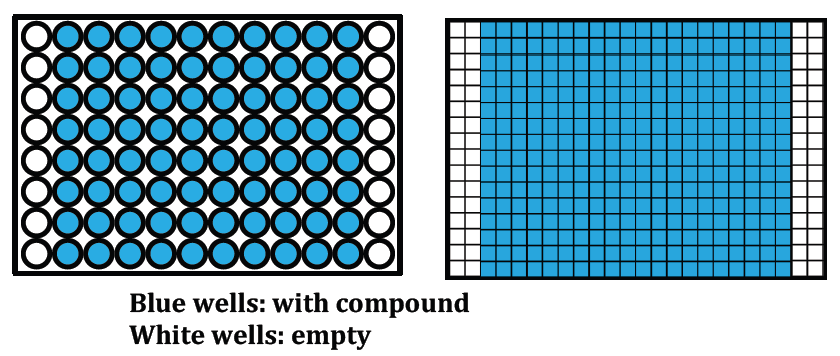
C. Volume of compound per well: 15 to 90 μl (384) or 50 to 200 μl (96)
D. Plates should be sealed with DMSO/low temperature-resistant,
removable (peelable) plate seals
3. Compound preparation:
Small molecules (synthetic or purified natural products) should be dissolved at 10 mM in net
DMSO (use DMSO of high quality and compatible with cell culture/molecular biology).
Natural products (polymers, fractions, extracts) can be dissolved at 100 mg/ml, ideally in neat
DMSO, at a concentration factor of 40x (for microbial broth extracts; or as recommended by the
sender, based on previous data.
4. Shipment:
Plates should be sealed as described above, reasonably cushioned to be protected from
mechanical shock and physical damage and shipped in dry ice.
Contact person and address for the shipment:
Prof. Lucio Freitas Junior
Departamento de Microbiologia
Instituto de Ciências Biomédicas
Universidade de São Paulo
Av. Professor Lineu Prestes, 1374
Edifício Biomédicas II, 1o andar, lab 178
CEP 05508-900
São Paulo – SP
luciofreitasjunior@gmail.com
(19) 99913-1968